DNA polimerase
A DNA polimerase ou ADN Polimerase é uma enzima que catalisa a polimerização do ácido desoxirribonucléico (ADN). A DNA polimerase é usada de duas maneiras pelas células. Em primeiro lugar, é utilizada para sintetizar uma nova cadeia de ADN a partir de uma cadeia de ADN antigo (ou fita molde). Antes de uma célula se dividir em duas células, ela deve reproduzir a informação genética para a célula nova. Em segundo lugar, é utilizada para reparar o ADN. O código genético pode ser corrompido durante a replicação ou por agentes mutagênicos externos que levam à proteínas disfuncionais e até mesmo a morte celular. A DNA polimerase contém um mecanismo de revisão e reparo de DNA que é usado para encontrar e corrigir os erros.[1]
Função
A DNA polimerase é utilizada na replicação do ADN, na reparação do ADN, na sequenciação de ADN e na Reacção em Cadeia da Polimerase. A função da polimerase de ADN é adicionar ela para sintetizar com a terminação 3' para formar uma nova fita de ADN. Para fazer a nova fita, a DNA polimerase precisa de um iniciador (primer), que é um segmento de fita simples curto de ADN. O primer é conhecido como uma cadeia de ADN ou ARN “arranque”, porque é um material de partida para a síntese formar a nova cadeia de ADN. Durante a replicação, erros podem ser feitos. A DNA polimerase corrige os erros, sintetizando uma nova fita de ADN. [2]
Síntese de ADN
A síntese do ADN (ou [a replicação do ADN]) é copiar fitas de ADN. Em outras palavras, ela copia a informação genética a partir do ADN. Este processo é importante. Quando o ADN faz um novo ADN, ele usa a síntese do ADN, que copia os materiais genéticos dos filamentos do modelo de ADN. Cada ADN é produzido dessa forma.
As cadeias condutoras (leading strands) e as cadeias em atraso ou cadeias lentas (lagging strands) são utilizadas na sintese de ADN. As cadeias principais são conhecidas como as cadeias de ADN. Eles estão apontando para a direção "direita" (5'->3') de uma maneira contínua. Na terminação 3', A DNA Polimerase pode ser sintetizada para construir um outro filamento devido ao grupo OH livre da terminação 3'.
As cadeias em atraso estão localizadas nos lados opostos das cadeias condutoras. Obviamente, elas apontam para a direção “esquerda” (3'->5'). Improvávelmente, a DNA polimerase não pode construir uma outra vertente na direção “esquerda” (3'->5'). Dessa forma, sintetizam com os fragmentos de Okazaki que estão fazendo as cadeias mais atrasadas na replicação. Após este processo, os primases são construídas. E, em seguida, a DNA polimerase pode utilizar o grupo OH livre 3' e sintetizar para construir uma outra fita na direção 5'-> 3'. [3]
Reparos de ADN
A revisão (proofreading) é um dos mecanismos de reparação do ADN com a DNA polimerase. À medida que a DNA polimerase adiciona novos nucleótidos pode cometer erros, que podem induzir mutações no código genético. A revisão verifica erros cometidos durante a replicação.(Purves, p227) Quando a revisão descobre erros, corrige-los através de um dos seguintes mecanismos. No entanto, por vezes, os erros não são reconhecidos durante a revisão, e pode continuar a ser uma parte permanente do genoma.(Purves, p253)
Reparo de pareamentos incorretos
A reparação de pareamentos incorretos (mismatch repair) é um mecanismo que verifica e corrige pares de bases com pareamento incompatível. Ela é diferente da revisão. A revisão verifica e corrige erros na replicação. Em contraste, a reparação de pareamentos incorretos verifica e corrige o DNA incompatível em relação ao emparelhamento de bases após a replicação.(Purves, p227)
Reparo por excisão
O reparo por excisão é um mecanismo que remove as bases anormais. Bases anormais podem ser formadas por danos químicos e substituir com bases anormais as bases normais (funcionais). O reparo de pareamento incorreto é utilizado para uma pequena porção de dano, mas a reparação por excisão é usada para grande parte dos danos que não podem ser reparados pelo reparo de pareamento incorreto. (Purves, p227)
Uso em Biotecnologia
Reação em Cadeia da Polimerase
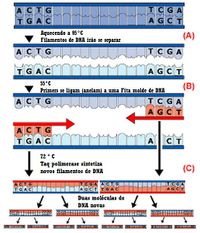
A Reação em Cadeia da Polimerase (Polymerase Chain Reaction - PCR) é uma técnica de laboratório que utiliza uma polimerase de ADN estável ao calor para fazer múltiplas cópias de ADN. Hoje em dia a PCR é uma técnica essencial no domínio da biotecnologia. A técnica envolve o uso de ciclos de temperatura repetidos e aumenta o número de ADN de curta região exponencialmente num curto espaço de tempo.
O processo cíclico envolve três etapas. Em primeiro lugar, a estrutura de hélice dupla de ADN de cadeia simples é separada em uma cadeia simples de ADN pelo calor. Um primer, uma fita de DNA “iniciadora” é adicionada, juntamente com a DNA polimerase estável pelo calor e quatro desoxirribonucleótido-trifosfatos, tais como dATP, dGTP, dCTP, e DTTP. Por último, quando resfriada a DNA polimerase copia os novos filamentos de ADN. O PCR requer seqüências de bases no final 3’ da fita de ADN e dois primers, com cerca de 15-20 bases de comprimento, ligando uma região do ADN.
A polimerase termoestável é necessária porque, para desnaturar o ADN, deve ser aquecida até 90°C, mas as polimerases de ADN são normalmente destruídas a esta temperatura. Thomas Brock descobriu pela primeira vez uma bactéria chamada Thermus aquaticus vivendo em fontes termais que podem viver em temperaturas de até 95°C, e cuja DNA polimerases não desnatura. Kerry Mullis percebeu as possíveis aplicações desta bactéria resistente ao calor, e acrescentou ela durante o processo cíclico. Por este trabalho, ele ganhou o Prêmio Nobel e a técnica teve grande impacto na biotecnologia. A DNA polimerase estável ao calor que foi isolada a partir do Thermus aquaticus e agora é usada em PCR é chamada Taq Polymerase.(Purves, p229-230)
Seqüenciamento de DNA
Além do PCR, Polymerase Chain Reaction (Reação em Cadeia da Polimerase), existe uma outra técnica importante para determinar a base da sequência de ADN. Esta técnica é a sequenciação de ADN. Seqüenciamento de DNA é o uso de nucleosídeos artificialmente alterados. Quatro letras, A, C, G, e T, está representando quatro subunidades de nucleotídeos de fita de ADN. A representa a adenina, C representa a citosina, G representa a guanina e T representa a timina. They are important subunits for the DNA sequencing, because they are carriers of genetic information. [4]
Para fazer o seqüenciamento de ADN, ele deve ter quatro substâncias para trabalhar com ele. Antes de usar a sequenciação de ADN, os fragmentos de ADN devem ser desnaturados. Após os fragmentos de ADN terem desnaturado, haverá apenas uma simples cadeias de ADN. E então, as cadeias simples serão colocadas em tubos de ensaio e misturados com quatro substâncias. As quatro substâncias são:
- A DNA polymerase que ajudará a sintetizar as novas cadeias,
- Primers artificiais que vão começar sintetizar as novas fitas,
- Quatro dNTP que são dATP, dGTP, dCTP, e dTTP, e
- Quatro ddNTP (Purves, p231)
Famílias de DNA Polimerases
DNA polimerases procarióticas
Nas bactérias, cinco tipos diferentes de DNA polimerase procariotas são encontrados.
- Pol I: Polimerase I foi descoberta por Arthur Kornberg, em 1958, e é conhecida como a DNA polimerase, ou polimerase de ADN. É polimerase útil para a reparação do ADN, porque tem tradução por cortes (5'->3') e revisão (3'->5').
- Pol II: Polimerase II é utilizada principalmente no reparo do ADN. É semelhante à polimerase I, porque tem tanto tradução por cortes (5'->3') e revisão (3'->5'). Em contraste com a polimerase I, a polimerase II, carece de atividade exonuclease 5'->3', e não pode usar um modelo duplex cortado.
- Pol III: Polimerase III é uma polimerase principal nas bactérias. Tem com uma capacidade de correcção (3'->5').
- Pol IV: Polimerase IV é uma família Y de polimerase de ADN.
- Pol V: Polimerase V é uma família Y de polimerase de ADN. [5]
DNA polimerases eucarióticas
Eucariotas tem pelo menos quinze polimerases de ADN. Estas cinco polimerases abaixo são as polimerases essenciais da DNA polimerase eucariótica.
- Pol α: Polimerase α é uma primase.
- Pol β: Polimerase β é usada na reparação do ADN.
- Pol γ: Polimerase γ replica o ADN.
- Pol δ: Polimerase δ é uma polimerase principal nas polimerases de ADN eucarióticos. Tem uma capacidade da correcção (3'->5').
- Pol ε: Polimerase ε é uma polimerase suplente da polimerase δ. [6]
História
Em 1959, Arthur Kornberg isolou a enzima DNA polymerase a partir de Escherichia coli.[1] Posteriormente, em 1959, Kornberg foi agraciado com o Prêmio Nobel de Fisiologia ou Medicina por seu trabalho.[2]
Referências
- ↑ Griffiths, Anthony J. F.; Wessler, Susan R.; Lewontin, Richard C.; Carroll, Sean B. Introdução à Genética. 9ª ed. Rio de Janeiro, RJ: Guanabara Koogan, 2009. p. 236-237. ISBN 978-85-277-1497-6
- ↑ The Nobel Prize in Physiology or Medicine 1959. Nobel Foundation. Página visitada em 20 de agosto de 2013.
Ligações externas
- Purves, Sadava, Orians, and Heller. Life: The Science of Biology. Virginia: Gordonsville, 2004
- DNA polymerase Wikipedia
- DNA polymerase Answers.com
- DNA replication Wikipedia
- DNA sequencing Wikipedia
Ver também
| ||||||||||||||