Splicing alternativo
Splicing alternativo, recomposição alternativa ou processamento alternativo, é um processo regulatório durante a expressão gênica, que resulta em um único gene codificando duas ou mais proteínas.[1] Isto é, o fenômeno pelo qual o mesmo gene dá origem a várias transcrições diferentes, dependendo de qual a combinação de éxons são incorporados no ARNm maduro.[2] Por razões que são até agora desconhecidas, a proporção de genes de splicing alternativo variam de acordo com cada espécie. Por exemplo o splicing alternativo é raro em plantas enquanto que em seres humanos mais de 70% dos genes são suscetíveis ao splicing alternativo.[3] Transcrições de splices alternativos podem ser identificadas para quase todo gene humano.[4] Devido ao de splice alternativo aproximadamente 30.000 genes humanos podem codificar para entre 64.000 a 96.000 proteínas diferentes.[1][nota 1] O gene humano normal contém entre 10 e 15 éxons e codifica três ou mais proteínas diferentes, devido a processamento alternativo.[2] O splicing alternativo pode ser constitutivo ou regulado. No primeiro caso, mais do que um produto é sempre produzido a partir do gene transcrito e no último caso, as diferentes formas são geradas em momentos diferentes, em condições diferentes, ou mesmo em células ou tipos de tecido diferentes.[5] Na maioria dos casos, as proteínas sintetizadas a partir de um gene por splicing alternativo são idênticas ao longo da maioria de seu comprimento mas diferindo em regiões-chave que podem afetar propriedades importantes, como a sua localização celular, a cinética de sua atividade catalítica ou os tipos de ligandos que podem se vincular.[6]
História
O splicing alternativo foi observado pela primeira vez no final de 1970, enquanto os bioquímicos estavam estudando o bacteriófago φX174.[7][8] Eles descobriram que o genoma deste bacteriófago dirigiu à produção de mais proteínas do que era esperado com base no tamanho do seu ADN. Eles resolveram este paradoxo demonstrando que alguns dos genes do φX174, na verdade, se sobrepõem.[7]
Splicing alternativo e íntrons
Os íntrons tornam o splicing alternativo possível, mas eles não são, aparentemente, apenas espaços biologicamente inertes. Há evidências crescentes de que eles desempenham muitas funções, incluindo a regulação do splicing alternativo.[9][10][nota 2] Proteínas que regulam o splicing se ligam a locais específicos chamados intensificadores de splicing exônicos (exonic splicing enhancers)(ESE) ou intensificadores de splicing intrônicos (intronic splicing enhancers)(ISE) que melhoram o splicing em sítios de splice próximos. Estas proteínas podem também ligar-se a outro tipo de locais específicos chamados silenciadores. Este sites são chamados de silenciadores de splicing exônicos (exonic splicing silencers)(ESS) ou silenciadores de splicing intrônicos (intronic splicing silencers) (ISS) e reprimem a splicing em em sítios de splice próximos.[5] Em genes com vários íntrons, alguns destes íntrons podem ser removidos separadamente ou em combinação dependendo como a maquinaria de recomposição interage com o ARN.[11]
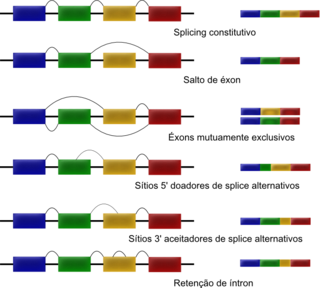
Tipos de eventos de splicing alternativo
- Salto de éxon ou cassete de éxons: Um éxon pode ser incluído ou ignorado (um cassete de éxons).[4]
- Éxons mutuamente exclusivos (ou excludentes): O ARNm maduro inclui ou o éxon em verde ou o éxon em amarelo na figura, mas não ambos, ou nenhum.
- Sítios 5' doadores de splice alternativos: Uma junção de splice 5' alternativa (sítio doador) é usada, alterando a fronteira a 3' do éxon a montante.
- Sítios 3' aceitadores de splice alternativos: Uma junção de splice 3' alternativa (sítio aceitador) é usada, alterando a fronteira a 5' do éxon a jusante.
- Retenção de íntron: Um íntron ou é retido ou excluído.
Veja também
Notas
- ↑ Este número é uma estimativa e pode variar. Por exemplo, outro autor fornece a estimativa de que nós, seres humanos, possuímos cerca de 25.000 genes e estes genes codificam mais do que cerca de 100.000 proteínas, como em Griffiths, Anthony J. F.; Wessler, Susan R.; Lewontin, Richard C.; Carroll, Sean B.. Introduction to Genetic Analysis. 9ª ed. New York: W. H. Freeman and Company, 2008. p. 296. ISBN 978-0-7167-6887-6.
- ↑ Após um debate entre Stephen Meyer, Stephen Matheson e Arthur Hunt na Biola University, uma guerra de blogs ocorreu incluindo Stephen Matheson, Richard Sternberg e Laurence Moran. Moran e Matheson tentaram ridicularizar a afirmação de Stephen Meyer de que muitos íntrons não são de fato DNA lixo alegando que apenas um punhado de íntrons tem função. Richard Sternberg respondeu a Matheson calculando que uma estimativa razoável para o número de íntrons humanos que sofrem splicing alternativo era de 90% de 190.000, ou seja, 171.000. Sternberg concluiu que, mesmo se a sua estimativa estivesse fora por um fator de dois, o número de íntrons funcionais seria ainda muito maior do que estimativa Matheson. No final da guerra de blogs Moran retificou que, pelo menos, 22.500 íntrons estavam envolvidos no splicing alternativo. Este número está bem acima dos supostos 1.000 íntrons. No entanto, este número ainda é bem inferior a evidência mostrada por Sternberg e com base em artigos revisados por pares como em Wells, Jonathan (8 de junho de 2010). The Fact-Free "Science" of Matheson, Hunt and Moran: Ridicule Instead of Reason, Authority Instead of Evidence. Evolution News and Views. Página visitada em 24 de fevereiro de 2013.
Referências
- ↑ 1,0 1,1 Hartl, Daniel L. Essential Genetics: A Genomics Perspective. 5ª ed. Sudbury, Massachusetts: Jones and Bartlett Publishers, 2011. p. 29; 318-319. ISBN 978-0-7637-7364-9
- ↑ 2,0 2,1 Gibson, Greg; Muse, Spencer V. A Primer of Genome Science. Sunderland, MA: Sinauer Associates, Inc., 2004. p. 94-96. ISBN 0-87893-232-1
- ↑ Griffiths, Anthony J. F.; Wessler, Susan R.; Lewontin, Richard C.; Carroll, Sean B.. Introduction to Genetic Analysis. 9ª ed. New York: W. H. Freeman and Company, 2008. p. 309. ISBN 978-0-7167-6887-6
- ↑ 4,0 4,1 Strachan, Tom; Read, Andrew P. Human Molecular Genetics. 4ª ed. New York: Garland Science, 2011. p. 373-374. ISBN 978-0-8153-4149-9
- ↑ 5,0 5,1 Watson, Jamed D.; Baker, Tania A.; Bell, Stephen P.; Gann, Alexandrer; Levine, Michael. Losick, Richard. Molecular Biology of the Gene. 5ª ed. San Francisco, CA: Pearson, Benjamin Cummings/CSHL Press, 2004. p. 394-397. ISBN 0-8053-4642-2
- ↑ Karp, Gerald. Cell and Molecular Biology: Concepts and experiments. 5ª ed. Hoboken, NJ: John Wiley & Sons, 2008. p. 531. ISBN 978-0-470-04217-5
- ↑ 7,0 7,1 Rana, Fazale. The Cell´s Design: How Chemistry Reveals the Creator´s Artistry. Grand Rapids, Michigan: Baker Books, 2008. p. 151-153. ISBN 978-0-8010-6827-0
- ↑ Berget S. M.; Moore C.; Sharp P. A.. . "Spliced segments at the 5' terminus of adenovirus 2 late mRNA" 74. DOI:10.1073/pnas.74.8.3171. PMID 269380. Bibcode: 1977PNAS...74.3171B.
- ↑ Wells, Jonathan. The Myth of Junk DNA. Seattle: Discovery Institute Press, 2011. p. 40. ISBN 978-1-9365990-0-4
- ↑ Meyer, Stephen C. Signature in the Cell. New York: Harper One, 2009. p. 407. ISBN 9780061472787
- ↑ Snustad, D. Peter; Simmons, Michael J.. Fundamentos de Genética. 4ª ed. Rio de Janeiro: Guanabara Koogan, 2008. p. 628. ISBN 978-85-277-1374-0
Ligações externas
| |||||||||||||||||