Separação incompleta de linhagens
Separação incompleta de linhagens ou segregação incompleta de linhagens, classificação incompleta de linhagens ou ainda coalescência profunda descreve um fenômeno onde a árvore filogenética produzida por um único gene difere da árvore em nível de população ou espécie, produzindo uma árvore discordante. O paradigma de SLI é caracterizado por segmentos de DNA que produzem árvores filogenéticas com diferentes topologias em comparação com árvores evolutivas hipotéticas inferidas.[1] Em outras palavras, na árvore de espécies, os dois parentes mais próximos não têm os dois alelos mais próximos. Em vez disso, eles têm alelos mais distantemente relacionados. A classificação de linhagem incompleta pode causar sérias dificuldades para a inferência filogenética.[2][3] Jeffrey Tomkins afirma:
| “ | A classificação de linhagem incompleta (ILS) é um daqueles segredos perpetuamente confusos no campo da biologia que tem atormentado o dogma fictício da evolução darwiniana por muitos anos.[4] | ” |
Exemplos
O pseudogene humano GULO
O gene GULO (L-gulonolactone oxidase) foi detectado em humanos, macacos, porquinhos-da-índia, morcegos, camundongos, ratos, porcos e aves passeriformes.[5] A perda de função do gene GULO afeta principalmente a capacidade das células de produzir vitamina C e foi detectada nos grandes símios e nos humanos, dentre outros grupos de seres vivos. Tomkins conduziu uma análise comparativa dos genes GULO humano e de macacos e os resultados podem ser vistos na tabela abaixo:
| A identidade da sequência de DNA para cada exon do gene GULO humano em comparação com cinco táxons de macacos diferentes (sequências de DNA extraídas do navegador do genoma UCSC). As identidades incluem inserções e exclusões. | ||||||
|---|---|---|---|---|---|---|
| Identidade de sequência percentual em comparação com a humana | ||||||
| Exon do gene GULO humano | Comprimento (bases) | Chimpanzé | Gorila | Orangotango | Babuíno | Macaco Rhesus |
| 1 | 101 | 99.1 | 100 | 97.1 | 91.1 | 91.1 |
| 2 | 96 | 98.0 | 98.0 | 94.8 | 92.8 | 88.6 |
| 3 | 107 | 98.2 | 85.0 | 98.2 | 74.8 | 74.8 |
| 4 | 151 | 99.4 | 90.8 | 97.4 | 94.1 | 92.8 |
| 5 | 164 | 97.6 | 97.0 | 94.6 | 91.5 | 90.3 |
| 6 | 129 | 97.7 | 98.5 | 99.3 | 95.4 | 93.8 |
| Esta tabela: Autor: Jeffrey P. Tomkins. Proprietário dos direitos autorais: Answers in Genesis. Fonte de publicação: Answers Research Journal e o seu site, www.answersresearchjournal.org[5] | ||||||
De acordo com Tomkins, o gorila é consideravelmente mais semelhante ao humano nessa região do que o chimpanzé - negando a ordem inferida de filogenia. Essa tendência evolutivamente discordante generalizada observada ao construir árvores baseadas em sequência, não apenas entre humanos e macacos, mas em todo o espectro da vida eucariótica, é conhecida como separação incompleta de linhagens.[5]
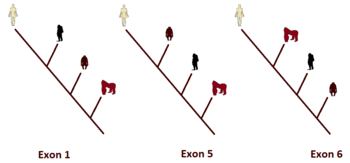
Olhando para a tabela de identidade de sequência de DNA, podemos construir uma árvore de similaridade de DNA simples, onde encontraremos uma tendência discordante, dependendo de qual exon usaremos para construir a árvore. Usando o exon 1, o gorila seria o mais próximo possível do homem; usando o exon 2, o gorila e o chimpanzé estariam empatados em segundo lugar; usando o exon 3, é o chimpanzé e o orangotango que estariam empatados como o macaco mais próximo; usando o exon 4, o gorila estaria mais longe dos babuínos e dos macacos Rhesus!; usando o exon 6, o orangotango seria o macaco mais próximo do ser humano. Apenas o exon 5 traria a filogenia esperada pelos evolucionistas. Na figura ao lado, as árvores filogenéticas dos exons 1, 5 e 6 são desenhadas para comparação.
A filogenia Bovini e o Wisent
O bisão (Bison bonasus) ou bisão europeu e o bisão americano são morfologicamente semelhantes e com cruzamento fértil entre si e têm genes nucleares intimamente relacionados, mas a filogenia do mtDNA agrupa consistentemente o bisão com a linhagem que leva ao gado taurino e zebu. Uma análise coalescente indicou que a discordância de filogenia poderia ser bem explicada por ILS apenas, de acordo com Kun Wang et al.[6]
Referências
- ↑ Tomkins, Jeffrey; Bergman, Jerry. (Dezembro 2013). "Incomplete lineage sorting and other ‘rogue’ data fell the tree of life". Journal of Creation 27 (3): 84–92.
- ↑ Maddison, Wayne P; Knowles, L Lacey. (Fevereiro 2006). "Inferring Phylogeny Despite Incomplete Lineage Sorting". Systematic Biology 55 (1): 21–30.
- ↑ Futuyma, Douglas J.. Evolution. Sunderland, Massachusetts: Sinauer, 2005. p. 38. ISBN 978-0-87893-187-3
- ↑ Tomkins, Jeffrey. More Than a Monkey: The Human-Chimp DNA Similarity Myth. [S.l.]: CreateSpace Independent Publishing Platform, 2012. p. 72. ISBN 978-147525325-2
- ↑ 5,0 5,1 5,2 Tomkins, Jeffrey. The Human GULO Pseudogene—Evidence for Evolutionary Discontinuity and Genetic Entropy. answersingenesis.org. Página visitada em 2020-09-21.
- ↑ Wang, Kun; Lenstra, Johannes A.; Liu, Liang; Hu, Quanjun; Ma, Tao; Qiu, Qiang; Liu, Jianquan. (19 de outubro de 2018). "Incomplete lineage sorting rather than hybridization explains the inconsistent phylogeny of the wisent". Communications Biology 1 (169).
External links
| |||||||||||||||||