Reação em cadeia da polimerase
A Reação em Cadeia da Polimerase (em inglês Polymerase Chain Reaction - PCR) é uma técnica para replicar uma seção específica do ADN e o amplificar exponencialmente.[1] Ela foi concebida pela primeira vez por Kary Mullis em 1983,[2] que teve a idéia ao pensar em uma maneira de analisar mutações no ADN. Posteriormente, em 1993, lhe foi atribuído o Prêmio Nobel de Química pelo seu trabalho.[2][3] Mais tarde, ele disse em uma revista científica, "Começando com uma única molécula de ADN de material genético, o PCR pode gerar 100 mil milhões de moléculas semelhantes em uma tarde. A reação é de fácil execução. Ela não requer mais do que um tubo de ensaio, alguns reagentes simples, e uma fonte de calor."[1]
Requerimentos
Para que o PCR possa funcionar, existem alguns requisitos que são necessários. Em primeiro lugar, é necessário um molde de ADN com a região desejada de ADN que irá ser amplificada. Em segundo lugar, tem de haver um ou mais primers ou iniciadores, que designam a zona de ADN que será amplificada por PCR. Em terceiro lugar, se precisa de uma DNA polimerase com uma temperatura ideal em torno de 70oC, que irá sintetizar uma cópia do fragmento de ADN. Em quarto lugar, é necessária os quatro trifosfatos desoxirribonucleotídeos dATP, dGTP, dCTP, e dTTP, que são os blocos de construção do novo ADN.[4] Em quinto lugar, é preciso uma solução tampão, que permite um ambiente adequado, sem contaminação. Finalmente, ele precisa de cátions bivalentes de íons de magnésio e de cátions monovalentes de íons de potássio.[2]
Primers
Sem primers, a PCR não seria capaz de amplificar uma seção específica do ADN. Os primers são normalmente muito curtos, usualmente não mais do que 25 pares de bases de comprimento, mas eles são necessários para a PCR trabalhar. Os primers são constituídos por nucleótidos que são complementares aos nucleótidos em cada extremidade do fragmento de ADN que irão ser amplificados. Isto significa que um nucleótido de adenina (A), vai emparelhar com um nucleotídeo timina (T), e um nucleótido citosina (C) vai emparelhar com um nucleótido guanina (G).
Taq Polymerase
A Taq Polymerase é uma DNA polymerase que foi isolada a partir de bactérias resistentes à temperatura Thermus aquaticus, e é usada durante a PCR para amplificar o fragmento de ADN.[1] A Taq é comumente utilizado em PCR, porque é capaz de resistir a temperaturas superiores a 70oC, o que é extremamente importante para que a reação possa trabalhar. A Taq catalisa a polimerização de ADN na direção 5' a 3'. Isto significa que não existe uma técnica de revisão disponível para ela, e a taxa de erro da Taq é 2.2 x 10-5 por nt por ciclo.[3] Isto é cerca de um erro para cada 10.000 nucleotídeos.[4]
Usos
A Reação em Cadeia da Polimerase é comumente utilizada para muitas coisas. É extremamente fácil de fazer, e requer muito pouco equipamento, mas é de vital importância para um monte de trabalho envolvendo ADN. Estas são apenas algumas das muitas técnicas de PCR para as quais é utilizada
Seqüenciamento de ADN
A sequenciação de ADN é utilizada para encontrar a ordem de nucleotídeos de um fragmento de ADN. Conhecer a seqüência dos nucleotídeos é muito importante para encontrar todas as doenças hereditárias, ou apenas para entender melhor o ADN. O PCR é utilizado para amplificar a seção de ADN que precisa ser sequenciada de modo a que as amostras suficientes estejam disponíveis. O ADN é então dividido em quatro amostras de cadeia simples separadas. Cada uma das amostras tem o seu próprio didesoxinucleótido (ddNTP) separado, que é marcado radioativamente de modo que ele pode ser visto mais tarde. Uma vez que as amostras são separadas usando electroforese em gel, elas são comparados lado a lado. O primeiro nucleótido é tanto A, T, C, quanto G, dependendo de qual ddNTP foi utilizado nessa amostra. Por exemplo, se a primeira tira no gel estava na amostra utilizando ddATP, então este nucleotídeo irá ser um ATP. Se a próxima tira do gel estava na amostra ddGTP, então este será o nucleotídeo GTP. Esta leitura de nucleotídeos é continuada para pela tira até que a sequência de nucleótidos completa daquela seção do ADN seja conhecida.[5]
Impressão digital genética
Esta é a técnica de identificação de algum indivíduo usando qualquer parte orgânica dele. Por exemplo, uma única gota de sangue ou um fio de cabelo pode ser usado, se eles estão em uma condição boa o suficiente. A PCR é utilizada neste processo para amplificar diferentes seções do DNA, que são, então, analisadas por meio de electroforese em gel. Equipes forenses muitas vezes usam essa técnica em cenas de crime para encontrar suspeitos.[6]
Clonagem de genes
A PCR é utilizada quando se faz a clonagem de genes para amplificar o gene, o qual é então inserido num vector. A clonagem de genes é muito diferente da clonagem de um organismo vivo, tal como uma ovelha. Em vez disso, é o processo de isolamento de um gene de um organismo e sua transferência para outro. Isto é muito útil para estudar os efeitos de um único gene, em um outro organismo.[7]
Procedimento
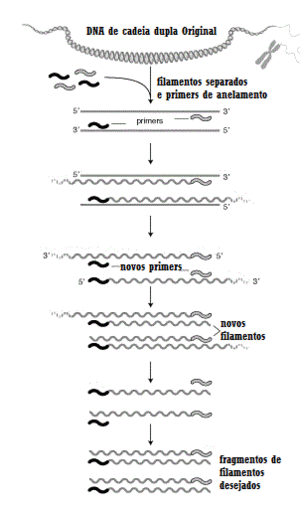
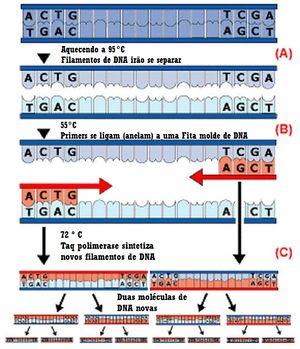
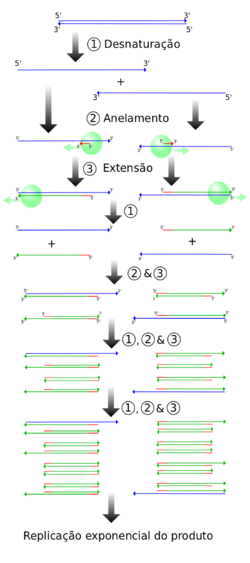
A PCR tem três etapas principais, as quais são repetidas em qualquer lugar de 20 a 40 vezes. Os três passos são:
1. Desnaturação - A cadeia de ADN é aquecida a cerca de 94oC, e divide-se para além de se tornar uma cadeia simples de ADN. O calor quebra as ligações de hidrogênio que estão mantendo as fitas de ADN juntas.
2. Anelamento - Os primers e seus nucleotídeos complementares na fita única de ADN formam e quebram ligações iônicas até os laços tornarem-se fortes o suficiente para não quebrar.
3. Extensão - Durante este passo, a temperatura é aquecida até cerca de 72oC, e a Taq polymerase polimeriza as bases que se formaram sobre o fragmento de ADN. A polimerização adiciona nucleótidos à extremidade 3' de cada primer que está ligado com a cadeia de ADN. Esta forma novas cadeias de ADN que contêm a secção de ADN que foi originalmente desejada.
Estes passos são repetidos tantas vezes o suficiente de modo a que os fragmentos são obtidos para ser utilizados na sequenciação do ADN e replicação de ADN.[8]
Referências
- ↑ 1,0 1,1 Hartl, Daniel L. Princípios de Genética de População. 3ª ed. São Paulo: Funpec Editora, 2008. p. 10. ISBN 978-85-7747-022-8
- ↑ 2,0 2,1 Bartlett, J. M. S.; Stirling, D.. Em: Bartlett, J. M. S.; Stirling, D.. PCR Protocols 226. 2ª ed. Totowa, New Jersey: Humana Press, 2003. Capítulo: A Short History of the Polymerase Chain Reaction, p. 3-6. ISBN 1-59259-384-4
- ↑ The Nobel Prize in Chemistry 1993. Nobel Foundation. Página visitada em 20 de agosto de 2013.
- ↑ Griffiths, Anthony J. F.; Wessler, Susan R.; Lewontin, Richard C.; Carroll, Sean B. Introdução à Genética. 9ª ed. Rio de Janeiro, RJ: Guanabara Koogan, 2009. p. 622. ISBN 978-85-277-1497-6
Ligações externas
- PCR Wikipedia
- Taq Polymerase Wikipedia
- DNA Sequencing Wikipedia
- Taq Fermentas
- Principles of PCR University of Ghent
- Cloning DNA in the Test Tube Biology Pages
- PCR Animation Cell Biology