Biologia molecular
A biologia molecular é o estudo da biologia a um nível molecular, incluindo a estrutura, função e composição de moléculas biologicamente importantes, tais como o ADN, o ARN e as proteínas.[1] É o ramo da biologia que lida com a formação, estrutura, e função das macromoléculas essenciais para a vida, tais como os ácidos nucleicos e proteínas, e especialmente com o seu papel na replicação das células e a transmissão de sua informação genética.[2]
Dogma central da biologia molecular
O dogma central da biologia molecular foi afirmado por Francis Crick em 1958[3] e re-afirmado em um artigo publicado na revista Nature, em agosto de 1970.[4] O dogma central da biologia molecular afirma que:
| “ | "O ADN atua como um modelo para se replicar, e também é transcrito em ARN, e o ARN é traduzido em proteína."[5] | ” |

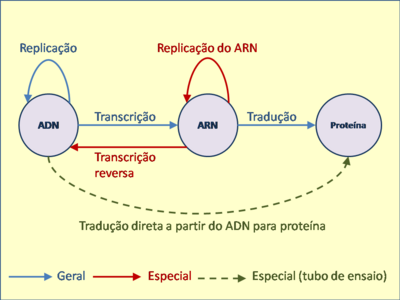
O dogma central afirma que uma vez que a "informação" passe para a proteína ela não pode passar adiante desta. A transferência de informação de proteína para proteína ou de proteínas para ácido nucleico é impossível. [7] Todas as células, desde a mais simples bactéria até os seres humanos, expressam sua informação genetica desta maneira - um principio fundamental deste dogma.[8] O autômato finito abaixo representa o dogma central:
Há casos especiais de transferência de informações biológicas seqüenciais. São estas a transcrição reversa, a replicação do ARN e a tradução direta a partir de ADN para a proteína. As transferências especiais foram indicadas no autômato acima em setas vermelhas. Em 1970, os primeiros relatórios na Nature forneceram a primeira evidência concreta para a existência da transferência de informação a partir de ARN para ADN em partículas de retrovírus, a transcrição reversa.[9] O impacto desta descoberta mudou de alguma forma o dogma central da biologia molecular aceito para o qual a transferência de informações é unidirecional. Esta descoberta levou à alteração do nome do vírus de tumor de ARN para retrovírus.[9] A replicação do ARN é a cópia de um ARN para outro. Muitos vírus se replicam dessa forma. As enzimas que copiam ARN para um novo ARN são chamadas ARN polimerases dependentes de ARN. Um vírus de ARN precisa de uma ARN polimerase dependente de ARN para transcrever os seus genes em ARNm.[10]
História
A biologia molecular foi criada na década de 1940 por pesquisadores inspiradas em conceitos metafóricos de código, linguagem e informação.[11] Em 1958, Francis Crick estabelece o dogma central da biologia molecular.[3]
Processos
Métodos

Sequenciamento de ADN
O sequenciamento de ADN é a determinação do total ou parte da sequência de nucleótidos de uma molécula específica de ADN.[12] Esta capacidade está no centro da revolução da biologia molecular.[12]
Eletroforese em gel
As moléculas de ADN linear são separadas de acordo com o tamanho, quando submetidas a um campo elétrico por meio de uma matriz de gel.[13] Após a electroforese se completar, as moléculas de ADN podem ser visualizadas por coloração do gel com corantes fluorescentes, como brometo de etídio.[13]
Copiando ADN: Reação em cadeia da polimerase (PCR)
Uma maneira de copiar o ADN em grandes quantidades é o método de PCR (reação em cadeia da polimerase). O PCR amplifica um fragmento de ADN curto e produz uma grande quantidade de cadeias idênticas.[14] A PCR de objetivo-único é uma técnica robusta e fiável para amplificar ácidos nucleicos mas ainda é sujeita a muitos artefatos e fatores ambientais.[15]
Southern blotting
Inventado e nomeado por Edwin Southern, este método é usado para sondar a presença de uma sequência específica de ADN no interior de uma amostra de ADN. É um método comum de usar sondas genéticas para olhar para as semelhanças ou diferenças, na estrutura de um gene.[16]
Northern blotting
É o procedimento mais simples para determinar se um gene é expresso numa amostra, mas ele realmente só confirma diferenças duplicadas ou maiores na expressão do gene uma vez que o método é semi-quantitativo.[17]
Western blotting
Western blotting, também denominado immunoblotting é um método para rastrear proteínas específicas em extratos livres de células. As proteínas são separadas primeiro de acordo com o tamanho pela técnica SDS-PAGE. As proteínas fracionadas são transferidas para uma folha de uma membrana de nitrocelulose e em seguida expostas a um anticorpo específico.[18] Muitas vezes, os anticorpos são marcados com enzimas para permitir a detecção.
Ligações externas
- Técnicas Básicas de Biologia Molecular por Diamar Costa-Pinto (FIOCRUZ)
Referências
- ↑ Definition of Molecular biology. Página visitada em 19 de Agosto de 2012.
- ↑ molecular biology. Página visitada em 19 de Agosto de 2012.
- ↑ 3,0 3,1 Crick, F.H.C.. (1958). "On Protein Synthesis". Symp. Soc. Exp. Biol. XII: 139-163.
- ↑ Crick, F.H.C.. (1970). "Central dogma of molecular biology". Nature 227 (5258): 561–3. DOI:10.1038/227561a0. PMID 4913914. Bibcode: 1970Natur.227..561C.
- ↑ Gibas, Cynthia; Jambeck, Per. Desenvolvendo Bioinformática. Rio de Janeiro: Editora Campus, 2001. p. 22. ISBN 85-352-0923-9
- ↑ Bolsover, Stephen R.; Hyams, Jeremy S.; Shephard, Elizabeth A.; White, Hugh A.; Wiedemann, Claudia G. Cell Biology (em inglês). Hoboken, New Jersey: John Wiley & Sons, 2004. 531 p. p. 39. ISBN 0-471-26393-1
- ↑ Waterman, Michael S. Introduction to Computational Biology (em inglês). Boca Raton, Florida: Chapman & Hall/CRC Press, 2000. p. 7. ISBN 0-412-99391-0
- ↑ Alberts, Bruce; Johnson, Alexander; Lewis, Julian; Raff, Martin; Roberts, Keith; Walter, Peter. Biologia Molecular da Célula. 5 ed. Porto Alegre: Artmed, 2010. p. 331. ISBN 978-85-363-2066-3
- ↑ 9,0 9,1 Flint, S. J.; Enquist, L. W.; Racaniello, V. R.; Skalka, A. M. Principles of Virology: Molecular Biology, Pathogenesis, and Control of Animal Viruses (em inglês). 2ª ed. Washington, D.C.: ASM Press. 217-218 p. p. 502. ISBN 1-55581-259-7
- ↑ Saunders, Venetia A.; Carter, John. Virology: principles and applications. Chichester: John Wiley & Sons, 2007. p. 74-75. ISBN 978-0-470-02387-7
- ↑ In: Konopka, Andrzej K.; Crabbe, M. James. Compact Handbook of Computational Biology. New York: Marcel Dekker, 2004. p. 4. ISBN 0-8247-0982-9
- ↑ 12,0 12,1 Alphey, Luke. DNA Sequencing:From Experimental Methods to Bioinformatics (em inglês). New York: Springer/BIOS Scientific Publishers, 1997. p. 1. ISBN 0-387-91509-5
- ↑ 13,0 13,1 Watson, James D.; Baker, Tania A.; Bell, Stephen P.; Gann, Alexander; Levine, Michael; Losick, Richard. Molecular Biology of the Gene. 5ª ed. San Francisco, CA: Pearson, Benjamin Cummings, 2004. p. 648. ISBN 0-8053-4635-X
- ↑ Pevzner, Pavel A.. Computational Molecular Biology: An Algorithmic Approach. Cambridge, Massachusetts: The MIT Press, 2000. p. 272-273. ISBN 0-262-16197-4
- ↑ Santalucia Jr., John. In: Yuriev, Anton. PCR Primer Design. Totowa, New Jersey: Humana Press, 2007. Capítulo: 1 - Physical Priciples and Visual-OMP Software for Optimal PCR Design, p. 3-14. ISBN 978-1-58829-725-9
- ↑ Dale, Jeremy W.; Park, Simon F. Molecular Genetics of Bacteria. 4ª ed. San Francisco, CA: John Wiley & Sons, 2004. p. 64-66. ISBN 0-470-85084-1
- ↑ Gibson, Greg; Muse, Spencer V. A Primer of Genome Science. Sunderland, MA: Sinauer Associates, Inc., 2004. p. 239-240. ISBN 0-87893-232-1
- ↑ Strachan, Tom; Read, Andrew. Human Molecular Genetics. 4ª ed. New York: Garland Science, 2011. p. 242-243. ISBN 978-0-815-34149-9